Cette page a été traduite automatiquement.
Merci de bien vouloir compléter un sondage de 1 minute concernant la qualité de cette traduction.
Développement d'un modèle de Deep Learning pour assister le diagnostic de pathologie
Par Ong Kok Haur, Laurent Gole, Huo Xinmi, Li Longjie, Lu Haoda, Yuen Cheng Xiang, Aisha Peng et Yu Weimiao, Bioinformatics Institute
Deuxième forme de maladie la plus courante chez les hommes, le cancer de la prostate est généralement diagnostiqué via l'inspection d'échantillons de tissus. Ce processus, traditionnellement réalisé par des pathologistes experts à l’aide d’un microscope, demande beaucoup de travail et prend du temps. En outre, le nombre ou la disponibilité de professionnels de santé, tels que les pathologistes, capables de réaliser de telles inspections, sont limités dans de nombreux pays, en particulier lorsque la charge de travail clinique est élevée. Cela peut entraîner des retards dans le traitement des échantillons à analyser et dans le démarrage du traitement.
Les recherches sur l’utilisation de l’IA et du Deep Learning pour aider au diagnostic pathologique du cancer de la prostate et d’autres formes de cancer se sont rapidement développées, en partie en raison des limites de l’analyse manuelle des échantillons. Plusieurs obstacles techniques doivent toutefois être surmontés avant que les modèles de Deep Learning puissent être développés, optimisés, validés et déployés pour des applications cliniques. Par exemple, on estime qu’environ 15 % des images numériques de pathologie présentent des problèmes de qualité liés à la mise au point, à la saturation et aux artefacts, entre autres problèmes. De plus, la qualité de l’image ne peut pas être évaluée quantitativement à l’œil nu, et les scanners d’images de diapositives entières (WSI) utilisés aujourd’hui produisent des ensembles de données extrêmement volumineux, ce qui peut compliquer le process de traitement d’images dans le cas d’images haute résolution de 85 000 × 40 000 pixels ou plus. Tout comme le diagnostic pathologique manuel, le processus d’annotation des images nécessite beaucoup de temps de la part des pathologistes expérimentés. Ce processus rend difficile la mise en place d’une base de données de haute qualité d’images étiquetées pour entraîner un modèle de diagnostic précis.
Le laboratoire de pathologie numérique computationnelle (CDPL) du Bioinformatics Institute (BII) d'A*STAR a développé une plateforme d'automatisation basée sur le cloud qui répond à de nombreux défis associés au diagnostic de pathologie, assisté par Deep Learning, tout en réduisant la charge de travail des pathologistes en matière d'étiquetage des images et de diagnostics cliniques (Figure 1). Cette plateforme comprend A!MagQC, un outil d'évaluation de la qualité d'image entièrement automatisé développé dans MATLAB® avec Deep Learning Toolbox™ et Image Processing Toolbox™. La plateforme comprend également un modèle de classification de Deep Learning formé pour identifier les modèles de Gleason. Lors d'expériences menées avec des pathologistes locaux et étrangers, la plateforme a réduit le temps d'étiquetage des images de 60 % par rapport à l'annotation manuelle et à l'examen microscopique traditionnel, aidant les pathologistes à analyser les images 43 % plus rapidement tout en conservant la même précision que l'examen microscopique conventionnel.
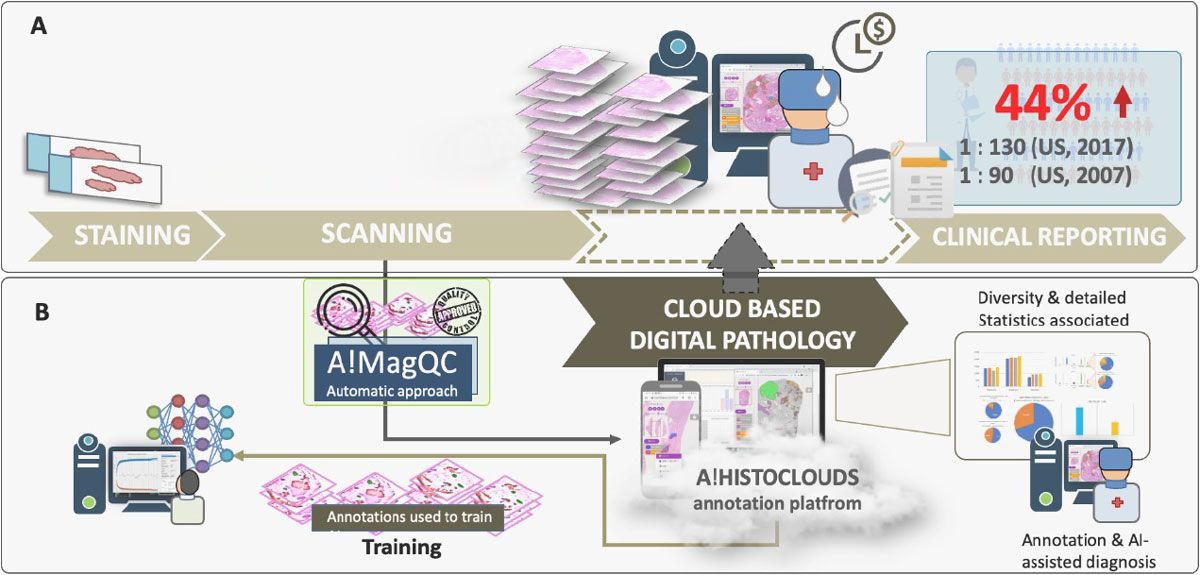
Figure 1. Plateforme d'analyse d'images numériques de pathologie, incluant A!MagQC et A!HistoClouds. A) montre le pipeline existant d’évaluation numérique de la pathologie. B) illustre le pipeline proposé dans cette étude, intégrant A!MagQC, A!HistoClouds et un modèle d'IA capable de détecter et d’évaluer le cancer de la prostate pour les images numérisées par plusieurs scanners dans le pipeline existant.
Évaluation de la qualité de l'image
En pathologie évaluée numériquement, les problèmes de qualité d'image peuvent être largement divisés en deux catégories : les problèmes de préparation d'échantillons de tissus et les problèmes de numérisation (Figure 2). Les déchirures tissulaires, les plis, les bulles d’air, les surcolorations et les sous-colorations entrent dans la première catégorie ; lorsque ces problèmes sont détectés et ont un impact sur le diagnostic, un nouvel échantillon devra être préparé. En revanche, lorsque des problèmes de contraste, de saturation et de mise au point de l’image sont détectés, l’échantillon existant peut simplement être rescanné et un nouveau prélèvement n’est pas nécessaire.

Figure 2. Uniformité de la texture, contraste, artefacts, saturation et problèmes de mise au point détectés avec A!MagQC. A) montre l'interface utilisateur simple et conviviale de A!MagQC. B) montre des exemples de fragments de mauvaise qualité à partir des images de diapositives entières. C) montre la sortie de A!MagQC où il est capable de représenter les zones de faible qualité d'une image de diapositive entière sous forme de cartes thermiques.
Que l’analyse soit effectuée par des pathologistes ou via des modèles de Deep Learning, chacun de ces problèmes courants peut avoir un effet néfaste. À ce titre, l’équipe BII CDPL d’A*STAR a développé des algorithmes de traitement d’image dans A!MagQC pour détecter automatiquement les principaux facteurs affectant la qualité de l’image. L’équipe a choisi MATLAB en raison des toolboxes spécialisées qu’il offrait. Par exemple, lorsque les images étaient trop volumineuses pour être chargées en mémoire, la fonction blockproc d'Image Processing Toolbox pouvait diviser chaque image en blocs d'une taille spécifiée, les traiter un bloc à la fois, puis assembler les résultats dans une image de sortie.
L’équipe a également utilisé les outils MATLAB pour créer l’interface utilisateur A!MagQC et pour compiler le code MATLAB dans un exécutable A!MagQC autonome pour la distribution.
À l’aide de la solution de contrôle qualité développée, l’équipe a quantifié la qualité de l’image pour identifier les variations de couleur, de luminosité et de contraste des WSI. Cet exercice a permis de garantir que le modèle de Deep Learning entraîné ultérieurement produirait des résultats de diagnostic précis pour la large gamme de scanners utilisés aujourd’hui.
Formation et test du modèle
Lors de l’analyse d’un échantillon, les pathologistes appliquent le système de classification de Gleason, spécifique au cancer de la prostate, pour attribuer un score en fonction de son apparence. Outre les tissus normaux ou bénins, les zones de l’échantillon peuvent inclure du stroma (tissu conjonctif) ou du tissu auquel est attribué un score de Gleason de 1 à 5, le score de 5 étant le plus malin (figure 3). Avant que l’équipe puisse commencer à entraîner un modèle de diagnostic d’IA pour classer les échantillons de tissus, elle devait assembler un ensemble de données de fragments d’images étiquetés avec ces catégories. Cette tâche a été réalisée avec l'aide de pathologistes utilisant A!HistoClouds, qui travaillaient avec des images dont la qualité avait été vérifiée à l'aide d'A!MagQC. Une fois que l’équipe disposait d’un ensemble de base de fragments d’images étiquetés, elle effectuait une augmentation des données pour étendre l’ensemble d’entraînement en reflétant les images individuelles verticalement ou horizontalement et en les faisant pivoter d’un nombre de degrés aléatoire ou ciblé.

Figure 3. Échantillons de tissus montrant du stroma, du tissu bénin et du tissu noté Gleason 3, Gleason 4 et Gleason 5. Les régions annotées (chacune étiquetée avec leurs catégories respectives) par les pathologistes dans A!HistoClouds seront extraites sous forme de fragments. Ces correctifs seront utilisés pour l’apprentissage du modèle.
En travaillant dans MATLAB avec Deep Learning Toolbox, l'équipe a créé des structures de modèles de Deep Learning en utilisant les réseaux pré-entraînés ResNet-50, VGG-16 et NasNet-Mobile, remplaçant leurs couches de classification habituelles par une couche de classification pondérée (Figure 4). L'équipe a également utilisé l’option multi-GPU pour passer d'un seul GPU à plusieurs GPU pour l’apprentissage du modèle de Deep Learning.
Le modèle est entraîné et appliqué via un processus itératif. Après la première étape d’apprentissage initiale sur des images étiquetées manuellement, vient une deuxième étape semi-automatisée au cours de laquelle les pathologistes examinent et modifient les prédictions générées par le modèle formé (Figure 5). Cette deuxième étape est répétée jusqu’à ce que le modèle soit prêt à être déployé par des professionnels de santé pour aider aux diagnostics cliniques. L’étape (a) nécessite une annotation manuelle initiale par les pathologistes de niveau junior et senior. Les annotations sont réalisées à l'aide d'A!HistoClouds, où elles sont extraites sous forme de correctifs utilisés pour entraîner le modèle de Deep Learning. Ce modèle générera ensuite la région d'intérêt (ROI) prédite pour aider les pathologistes, d'où le nom d'annotation semi-automatique. À l’étape (b), le modèle subira un apprentissage progressif, où les ROI prédits par l’IA seront examinés et corrigés par les pathologiste. Les ROI sont extraits sous forme de correctifs et le modèle apprend à partir de ces nouvelles données. L'étape (b) est répétée jusqu'à ce que les performances du modèle atteignent la convergence, où avec l'étape (c), le modèle sera déployé pour obtenir une annotation entièrement automatique/un diagnostic entièrement automatique qui aidera à la prise de décision des pathologistes.
Prochaines étapes
Depuis, le CDPL a déployé sa plateforme de diagnostic de pathologie assistée par Deep Learning sur des plateformes cloud mondiales, offrant un accès facilité à l’équipe de pathologistes travaillant dans différents pays. Le BII d'A*STAR travaille actuellement à la validation et à l'optimisation de son modèle de Deep Learning pour des scénarios cliniques supplémentaires, notamment avec différentes épaisseurs de tissus, d’autres mécanismes de coloration et scanners d'images. Enfin, BII explore les possibilités d’étendre le même processus d’évaluation de la qualité d’image et le même workflow de Deep Learning au-delà du cancer de la prostate, à d’autres types de cancer.
Le CDPL du BII a également organisé le Automated Gleason Grading Challenge 2022 (AGGC 2022), qui a été accepté par la International Conference on Medical Image Computing and Computer Assisted Intervention (Conférence internationale 2022 sur l'informatique d'imagerie médicale et l'intervention assistée par ordinateur). L'AGGC 2022 se concentre sur les défis liés à la classification de Gleason pour le cancer de la prostate, à l'exploitation numérique de la pathologie et aux approches de Deep Learning. Le défi vise à développer des algorithmes automatisés avec une grande précision pour des images histopathologiques de la prostate colorées par H&E présentant des variations dans le monde réel. Il s’agit notamment du défi inaugural dans le domaine de la pathologie numérique qui étudie les variations d’images et construit des modèles de diagnostic d’IA généralisables.
Bien que le défi soit relevé, l’ensemble complet des données est désormais disponible pour la poursuite de recherches.
Remerciements
Le BII de l'A*STAR souhaite remercier ses collègues de l'hôpital universitaire national (NUH), notamment le professeur Tan Soo Yong et le Dr Susan Hue Swee Shan, le Dr Lau Kah Weng et le Dr Tan Char Loo, etc. pour leur partenariat et leur collaboration. Le NUH est dûment reconnu comme étant à l’origine des données et des échantillons qui ont contribué à la recherche décrite dans ce travail. L’équipe est reconnaissante du soutien reçu d’autres partenaires cliniques et industriels.
Publié en 2024

